‘加拿大英属哥伦比亚大学(University of British Columbia)的团队意识到单细胞定序技术的限制,致力于革新定序流程,研发出直接样本库制备法(direct library preparation,DLP),省略全基因组增幅的步骤,排除增幅过程所造成的技术与人为偏误,而且比以往使用的方式更精准可信。’
单细胞定序 真实反映细胞的独特性
单细胞定序(single-cell sequencing)技术在 2013 年时,被《Nature Methods》期刊选为年度技术,这项技术突破了基因定序的应用,过去通常使用组织样本或细胞群进行 DNA 与 RNA 序列分析,透过分析运算样本细胞群取得平均值,但是这样的方式往往无法看到细胞个体的特性。
为了真实反映每个细胞的独特性,科学家分离出单一细胞,从中萃取遗传物质,再以全基因组增幅(whole-genome amplification,WGA)技术制造足量遗传物质以进行分析,单细胞基因体(single-cell genomics)被解码之后,更揭示了细胞个体的异质性(heterogeneity),应用于癌细胞的研究,可以更精确的了解单一癌细胞的基因体以及结构变化,更能侦测罕见的细胞变异,这项技术在其他领域如:微生物学、免疫学、神经生物学、胚胎发育学、组织嵌合(tissue mosaicism)以及产前诊断都有长足贡献。
精益求精 现行技术仍有限制
尽管单细胞定序的应用令人振奋,但仍有许多技术上的限制有待改良,其中以 WGA 为例,这是造成偏误的主要步骤之一,因为要符合现有的定序技术,科学家必须将单一细胞基因体复制放大,以产生足够的遗传物质制备分析样本库(library preparation),常见的 WGA 技术有:DOP-PCR(degenerate oligonucleotide-primed PCR)、MDA(multiple displacement amplification)以及 MALBAC(multiple annealing and looping based amplification Cycles),各种增幅技术都有适用的情况,但不论何种方式都很难避免技术偏差,对偶基因若有异型合子的突变(heterozygous mutation),该基因无法透过聚合反应增幅而造成对偶基因丢失(allelic dropout,ADO),变异基因的伪阴性或伪阳性,聚合酶链锁反应所使用的引子(primer)品质会影响增幅基因组的覆蓋率(coverage),覆蓋率不足、不一致的覆蓋率等偏误(图一)都使单细胞的基因序列无法被真实呈现。

图一 单细胞序列分析常见的偏误(a)对偶基因丢失、伪阳性或是伪阴性而造成覆蓋率不,其中 Pop 指细胞群体(a population of cell)(b)覆蓋率的度量包含覆蓋深度(coverage depth)以及总物理覆蓋率(total physical coverage),也就是覆蓋广度(coverage breadth)(c)覆蓋一致性(coverage uniformity)随着不同细胞而有差异。
UBC 团队的新技术 不需全基因组增幅
加拿大英属哥伦比亚大学的团队意识到 WGA 技术的限制,致力于革新单细胞定序的程序,在今年一月正式于《Nature Methods》发表新的研究成果,使用直接样本库制备法(direct library preparation,DLP),省略了 WGA 的步骤,不进行前置增幅(preamplification),排除基因增幅过程中所产生的技术与人为偏误,而且比以往使用的方式更精准可信。
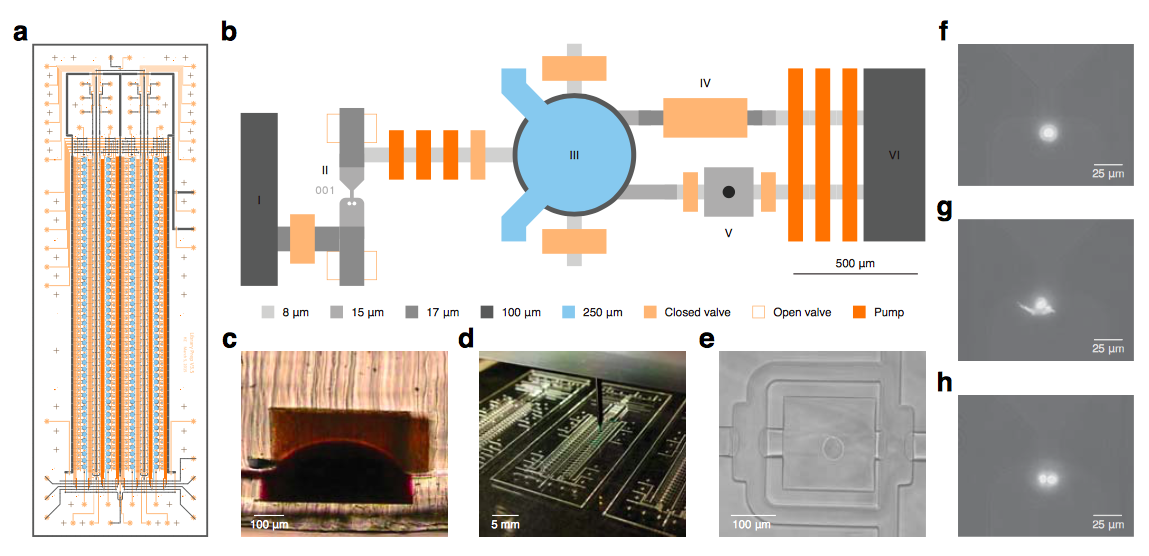
科学家设计了一组 192 孔的微流体装置(图二),这套系统使用奈生(nanoliter)作为单为容积,可以直接针对单一细胞进行样本库制备,除了利用极微量的容积提高分析的分辨率之外,可充气的微流体分析盘能够随着不同的实验需求而变化容积大小,同时还有内建的标记引子(incorporated index primers),可以直接标记单一细胞,此外独立的影像系统让研究人员能清楚的观察细胞样本。
技术前景无可限量
高分辨率的分析设备搭配改良的运算流程,研究团队分析癌细胞株,将个别单细胞定序结果以电脑模拟汇合,模拟大量基因体分析的结果,得出相当一致的覆蓋率,同时更精确的侦测癌细胞的基因复制数改变(copy-number alterations,CNAs),大幅提升单细胞异质性的侦测,相较于现行的技术,新技术的花费与旧法相当,但是科学家有机会从中获得更多资讯,让单细胞定序的应用更有价值。
研究的主持人 Carl Hansen 博士表示,已将新技术的专利注册并为 AbCellera 所有,AbCellera 是 Hansen 博士所领导的生技公司,主要致力单株抗体疗法(monoclonal antibody therapeutics),虽然 DLP 技术与 AbCellera 的发展关联性较低,Hansen 也因此规划成立新公司发展商品化 DLP,不过 Hansen 相信未来可以将技术延伸应用于免疫疗法,使更多人受惠于革新的技术。
文 / Joanne Shih
延伸阅读:美国拟严管基改动物 基因编辑的两难©www.geneonline.news. All rights reserved. 基因线上版权所有 未经授权不得转载。合作请联系:service@geneonlineasia.com
参考文献:
1.”Method Of The Year 2013″. Nature Methods 11.1 (2013): 1-1
2.Zahn, Hans et al. “Scalable Whole-Genome Single-Cell Library Preparation Without Preamplification”. Nature Methods 14.2 (2017): 167-173
3.Gawad, Charles, Winston Koh, and Stephen R. Quake. “Single-Cell Genome Sequencing: Current State Of The Science”. Nature Reviews Genetics 17.3 (2016): 175-188
4.Molika Ashford. “UBC group debuts single-cell sequencing method without whole genome amplification”. Genomeweb (2017)
5.Navin, Nicholas E. “Cancer Genomics: One Cell At A Time”. Genome Biology 15.8 (2014)
图片来源:
1.http://en.novogene.com/wp-content/uploads/2015/10/single-cell-RNA.png
2.Gawad, Charles, Winston Koh, and Stephen R. Quake. “Single-Cell Genome Sequencing: Current State Of The Science”. Nature Reviews Genetics 17.3 (2016): 175-188
3.Zahn, Hans et al. “Scalable Whole-Genome Single-Cell Library Preparation Without Preamplification”. Nature Methods 14.2 (2017): 167-173