在基因編輯中,CRISPR-Cas9 為當今最著名的技術之一。它比以前使用的技術更準確,並且具有廣泛的潛在應用,包含各種單基因遺傳性疾病治療。然而,要大量交付 CRISPR-Cas9 材料仍然很困難,以及切割 DNA 雙股的分子剪刀 Cas9 可能會切錯位點,導致脫靶效應,進而發生不可逆且不可預期的傷害。
近日,哈佛大學Wyss生物啟發工程研究所的研究團隊建立了一種新的基因編輯工具 Retron Library Recombineering(RLR)。RLR 使用細菌 DNA 片段 Retron,來產生單股 DNA 片段。它透過在細胞複製其基因體時引入另一種DNA來執行誘餌轉換,進而有效地產生基因變異而不會破壞 DNA。RLR 同時生成多達數百萬個變異,並「編碼」變異細胞,以便可以一次篩選整個文庫,進而可以輕鬆生成和分析大量數據,使科學家們同時進行數百萬個基因實驗。相關研究結果刊登於《Proceedings of the National Academy of Sciences》。
Retron Library Recombineering
由於 Retron 在細菌中有產生單股 DNA 的能力,該研究團隊開始研究其基因編輯功能。
他們為了確認能否透過 Retron 來完成有效重組,他們首先建立了細菌DNA的環狀質體,該質體包含位於 Retron 序列中的抗生素抗藥性基因,以及一個單股黏合蛋白(single-stranded annealing protein, SSAP)基因,可以將 Retron 序列整合入細菌基因組。他們將這些 Retron 質體插入到大腸桿菌(E. coli )中,以查看在 20 代細胞複製後該基因是否成功整合到其基因組中。最初,少於 0.1% 的帶有 Retron 重組系統的大腸桿菌摻入了所需的變異。
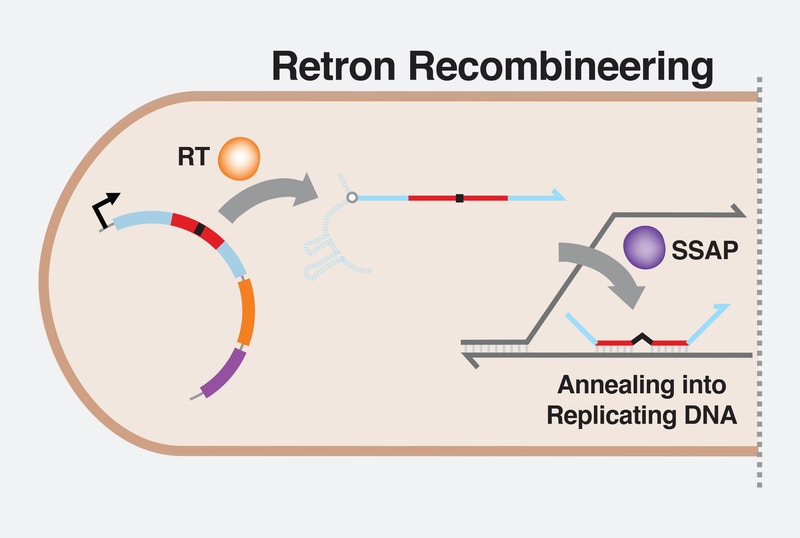
再來,為了改善起始狀況,他們對細菌進行了幾項基因調整。首先,他們使細胞的天然錯配修復機制失去活性,因此可以在將其遺傳給下一代之前固定所需的突變。他們還抑制了 2 個編碼外切核酸酶的細菌基因,外切核酸酶會破壞游離浮動的單股 DNA。這些變化極大地增加了整合 Retron 序列的細菌比例,達到了總數的 90% 以上。
既然確信其 Retron單股 DNA 已整合到細菌的基因體中,再來他們就測試了是否可以將 Retron當作基因定序的捷徑,進而可以在混合物中進行許多實驗。因為每個質體都有自己獨特的Retron,可以充當「名稱標籤」,他們認為它們應該能夠對短得多的 Retron 序列進行定序,而不是對整個細菌基因體進行測序,進而確定細胞已產生哪些突變。
他們也測試了 RLR 是否可以檢測到大腸桿菌中已知的抗生素抗藥性突變。他們發現,與其他突變相比,包含這些突變的序列在其定序數據中的存在比例要高得多。另外,他們還確定 RLR 具有足夠的靈敏度和準確度,可以量測由非常相似突變所引起的抗藥性的微小差異。此研究的關鍵點在於,透過對整個細菌文庫中的條形碼進行定序而不是對單一突變體進行分離和定序來收集這些數據,可以極大地加快這一過程。
再來,他們想知道 RLR 是否可以用於隨機片段化的DNA,並找出它們一次可以使用多少個 Retron。他們切碎了對另一種抗生素具有高度抵抗力的大腸桿菌菌株的基因體,並利用這些片段建立了包含在質體回溯序列中的數千萬條基因序列的文庫。然後,他們將該文庫引入 RLR 優化的大腸桿菌菌株中進行分析。他們再一次發現,通過對細菌庫進行定序,它們相對於其他細菌富集的事實可以很容易地辨識出賦予抗生素抗藥性的 Retron。
RLR 未來發展和應用
RLR 與 CRISPR 之間的另一個差異是,隨著細菌的複製,成功將所需突變整合到其基因體中的細菌比例會隨著時間的流逝而增加,而 CRISPR 的“單一”方法在首次嘗試時往往會成功或失敗。因此,RLR 可能與 CRISPR 結合使用以改善其編輯性能,或者可以取代一些 CRISPR 毒性系統。
然而,如何進一步提升 RLR 的精準度和標準化編輯率,都是未來急需努力的方向。
參考資料:
1. PNAS May 4, 2021 118 (18) e2018181118; https://doi.org/10.1073/pnas.2018181118
2. https://wyss.harvard.edu/news/move-over-crispr-the-retrons-are-coming/
©www.geneonline.news. All rights reserved. 基因線上版權所有 未經授權不得轉載。合作請聯繫:service@geneonlineasia.com