目前大多研究團隊主要利用質譜儀進行蛋白質定序,但該儀器仍有待改善之處,其一為靈敏度不高,它只能檢測到一百萬個拷貝數以上的蛋白,太低就無法檢測到。其二為低通量(throughput),它只能檢測單一樣本的數千種蛋白,因此需花較多資源和時間去篩檢所要的蛋白。
近日(10 月 22 日),由德洲大學奧斯汀分校(University of Texas at Austin)細胞和分子生物學研究所的Edward Marcotte 教授所率領的研究團隊開發出一種類似基因體次世代定序(next generation sequencing, NGS)的新技術來定序蛋白質,與現有的質譜儀技術相比,該技術有更高的靈敏度和通量,能獲得數百萬的胜肽分子定序結果,更能直接辨識單一蛋白分子,該研究發表於《Nature Biotechnology》期刊上。
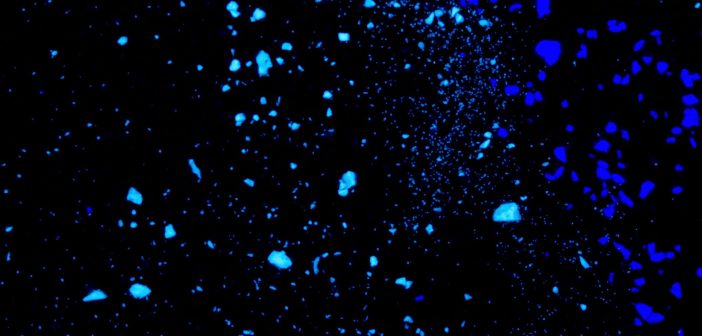
該研究團隊的 Angela Bardo 博士首先改造了一種用於 NGS 的成像技術,稱為 TIRF 顯微鏡,它能將化學標籤附加到待分析的蛋白質上。接著,他們利用該方法對單一樣本中的數百萬個蛋白分子進行定序。他們對多肽樣本中的半胱胺酸(cysteine)和離胺酸(lysine)殘基進行選擇性的螢光標記,然後將其固定在玻璃表面,接著在蛋白質經過連續 Edman 降解循環後,再透過全內反射顯微鏡方法(internal reflection microscopy)來監測每個一分子的螢光減少狀況。再來,他們將獲得的螢光序列分配到參考數據庫中的母體蛋白。
此外,他們也利用 zeptomole (10-21 mole) 級的合成胜肽和天然衍生胜肽測試了該新方法。同時,他們還對磷酸絲胺酸(serine)進行了螢光標記,實現了單分子中磷酸化位點的讀取。
Marcotte教授表示,若持續改進該技術,未來可檢測到單一樣本可望達到數十億的蛋白分子。該技術除了對生物醫學基礎研究產生重大影響,還更助於人們發現新的生物標記,以及協助癌症及其他疾病的診斷。例如,科學家有望能透過該技術分析每個細胞,以了解腫瘤是如何從一小部分相同的細胞發展成不同的細胞組,以及進一步分析每個細胞特性,進而發明新的方法或技術來攻擊癌症。
延伸閱讀:預測下一個生技世代的風貌(三): 蛋白質體學的即時掌握參考資料:
1. Nature Biotechnology, 2018; DOI: 10.1038/nbt.4278
2. https://news.utexas.edu/2018/10/22/new-protein-sequencer-could-transform-biological-research
©www.geneonline.news. All rights reserved. 基因線上版權所有 未經授權不得轉載。合作請聯繫:service@geneonlineasia.com