去年 7 月,张锋团队在《Science》发表一种单细胞核 RNA 定序的方法 (Single-nucleus RNA-sequencing, sNuc-seq) ,克服了以往鉴定冷冻或固定组织中基因表现的困难,如今相同团队针对 sNuc-Seq 低通量的阻碍,提出了突破性的解决方案──将 sNuc-Seq 结合微流体 (Microfluid)技术,使研究者得以在结构复杂的组织中大规模的鉴定单细胞之基因表现,此项名为 DroNc-Seq 的技术已于 8 月 28 日发表在《Nature Methods》。
揭开细胞层次的基因表现图谱
纵使在相同的组织或细胞种类中,各个细胞皆有其回应环境变化的独特方式,而这些“行为模式”决定于细胞基因表现的水平。针对细胞难以分离或大脑等复杂组织,传统的单细胞分析技术应用受限,且分离细胞所使用的酵素往往会破坏细胞结构,扭曲了原始的 RNA 讯息。
去年由张锋团队于《Science》所发表的研究利用 Div-Seq (结合 sNuc-Seq 与脉冲标定技术) 成功分析单一增殖细胞的基因表达状态,不仅能敏感地鉴定海马回中密切相关的细胞类型,亦能追踪新生神经元的转录动力学 (transcriptional dynamics),对于神经疾病的研究与治疗有莫大助益。核心研究者之一 Yinging Li 即表示,神经元细胞间的异质性是神经疾病难以发展有效疗法的原因之一,只有当全盘了解细胞层次的基因表现,才有机会揪出造成疾病的“凶手”并开发出有效的治疗方法。
DroNc-Seq:高通量的单细胞核 RNA 定序法
然而, sNuc-Seq 未能堪称完美,低通量的限制使其无法应用于成千上万个细胞核样本的研究,于是张锋团队将脑筋动到了微流体。Drop-Seq 为 2015 年发表于《Cell》的单细胞 RNA 测序技术,该技术系将单个细胞与带有独特 DNA 序列的磁珠 (DNA barcoded-beads) 包裹于微滴中,透过微滴的细胞裂解液使之释出 RNA,其可与磁珠上序列互补的 DNA 结合,随后经由乳化液回收微滴中的磁珠再行定序,此技术不仅扩大了单细胞基因表现的分析效能,亦大幅降低成本。受到 Drop-Seq 的启发,张锋团队融合了 sNuc-Seq 与微流体控制技术,利用分离组织的细胞核直接与带有 DNA 的磁珠结合后再定序,该技术命名为 DroNc-Seq。
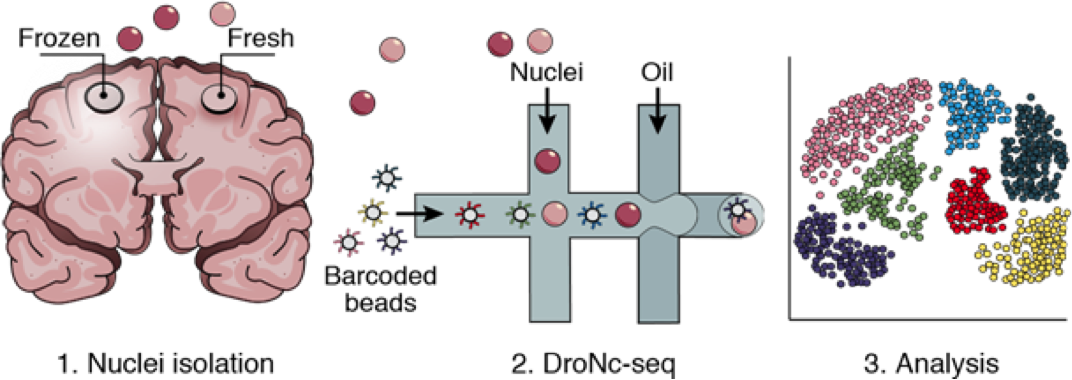 (注:图片取自《Nature Method》期刊,doi:10.1038/nmeth.4407)
(注:图片取自《Nature Method》期刊,doi:10.1038/nmeth.4407)
为了验证 DroNc-Seq 定序的准确度与效能,张锋团队利用小鼠细胞与取自 Genotype-Tissue Expression (GTEx) Project 的人类脑组织作为样本,同时与 Drop-Seq、sNuc-Seq 与其他低通量 scRNAseq 技术相行比较,发现 DroNc-Seq 的定序结果皆与先前研究结果一致,且能鉴别神经元、神经胶细胞与其他脑组织罕见细胞特有的表达特征,以及区分密切相关的细胞亚型,不论是检测的可靠度、灵敏度、规模或成本,皆具有高度竞争力。
开启疾病研究的新页
第一作者 Naomi Habib 表示, DroNc-Seq 可用于检测人体细胞亚群极微小的差异,并帮助研究者发现更多未知的稀有细胞,对于神经退化性疾病如阿兹海默症、癌症及正常脑部老化过程的研究皆有应用价值。此外,通讯作者之一的 Aviv Regev 已着手规划建立人类细胞图谱 (Human Cell Atlas), DroNc-Seq 将可能成为此计画主要应用的技术之一。
张锋团队已就 DroNc-Seq 的技术内容申请专利,且有数间公司表态未来授权的意愿,开发单细胞定序应用平台的英国生技公司 Dolomite Bio 将于近期发表 DroNc-Seq 的检测芯片,其微滴设计更小,而可增加高通量细胞核 RNA 定序的效能、成本更低,虽然总RNA 捕捉效率低于单细胞定序技术,却能提供更完整的细胞核 RNA 及 pre-mRNA 表达讯息。
单细胞定序于 2013 年被《Nature Method》列为年度技术,随着该技术的普及与成熟,研究者得更精确地由细胞层次探讨生物体中各种细胞种系的演化亲缘关系,在肿瘤基因体学、神经医学或发育生物学等研究领域皆开始发挥强大的应用潜力,根据美国透明市场研究公司 (Transparency Market Research) 的报告显示,全球转录体技术市场将于 2019 年达到 46 亿美金。过去的疾病研究大多聚焦在分子机制之探讨,期待透过高通量的单细胞核 RNA 定序技术,未来能以生态系的观点看待细胞与疾病之间的关系,开启临床医学研究的新纪元。
文 / Angela Chang
资料来源:
1. https://www.genomeweb.com/sequencing/advances-single-cell-rna-sequencing-enable-analysis-nuclei-higher-throughput
2, http://www.nature.com/nmeth/journal/v11/n1/full/nmeth.2801.html
3. https://www.broadinstitute.org/news/dronc-seq-microfluidic-single-nucleus-rna-sequencing
4. http://www.genengnews.com/gen-news-highlights/single-nucleus-rna-seq-merges-with-microfluidics/81254868
5. https://www.broadinstitute.org/blog/divide-and-conquer-new-single-cell-approach-broadens-range-cell-types-can-be-studied-brain
6. Habib, N. et al. Nature Methods (2017) doi:10.1038/nmeth.4407
7. Habib, N. et al. Science 353: 925–928 (2016).
©www.geneonline.news. All rights reserved. 基因线上版权所有 未经授权不得转载。合作请联系:service@geneonlineasia.com






