去年 7 月,張鋒團隊在《Science》發表一種單細胞核 RNA 定序的方法 (Single-nucleus RNA-sequencing, sNuc-seq) ,克服了以往鑑定冷凍或固定組織中基因表現的困難,如今相同團隊針對 sNuc-Seq 低通量的阻礙,提出了突破性的解決方案──將 sNuc-Seq 結合微流體 (Microfluid)技術,使研究者得以在結構複雜的組織中大規模的鑑定單細胞之基因表現,此項名為 DroNc-Seq 的技術已於 8 月 28 日發表在《Nature Methods》。
揭開細胞層次的基因表現圖譜
縱使在相同的組織或細胞種類中,各個細胞皆有其回應環境變化的獨特方式,而這些「行為模式」決定於細胞基因表現的水平。針對細胞難以分離或大腦等複雜組織,傳統的單細胞分析技術應用受限,且分離細胞所使用的酵素往往會破壞細胞結構,扭曲了原始的 RNA 訊息。
去年由張鋒團隊於《Science》所發表的研究利用 Div-Seq (結合 sNuc-Seq 與脈衝標定技術) 成功分析單一增殖細胞的基因表達狀態,不僅能敏感地鑑定海馬迴中密切相關的細胞類型,亦能追蹤新生神經元的轉錄動力學 (transcriptional dynamics),對於神經疾病的研究與治療有莫大助益。核心研究者之一 Yinging Li 即表示,神經元細胞間的異質性是神經疾病難以發展有效療法的原因之一,只有當全盤了解細胞層次的基因表現,才有機會揪出造成疾病的「兇手」並開發出有效的治療方法。
DroNc-Seq:高通量的單細胞核 RNA 定序法
然而, sNuc-Seq 未能堪稱完美,低通量的限制使其無法應用於成千上萬個細胞核樣本的研究,於是張鋒團隊將腦筋動到了微流體。Drop-Seq 為 2015 年發表於《Cell》的單細胞 RNA 測序技術,該技術係將單個細胞與帶有獨特 DNA 序列的磁珠 (DNA barcoded-beads) 包裹於微滴中,透過微滴的細胞裂解液使之釋出 RNA,其可與磁珠上序列互補的 DNA 結合,隨後經由乳化液回收微滴中的磁珠再行定序,此技術不僅擴大了單細胞基因表現的分析效能,亦大幅降低成本。受到 Drop-Seq 的啟發,張鋒團隊融合了 sNuc-Seq 與微流體控制技術,利用分離組織的細胞核直接與帶有 DNA 的磁珠結合後再定序,該技術命名為 DroNc-Seq。
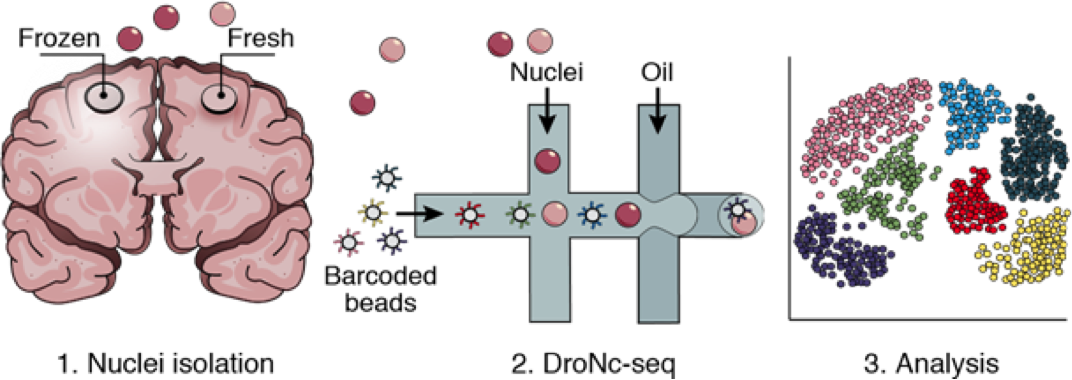 (註:圖片取自《Nature Method》期刊,doi:10.1038/nmeth.4407)
(註:圖片取自《Nature Method》期刊,doi:10.1038/nmeth.4407)
為了驗證 DroNc-Seq 定序的準確度與效能,張鋒團隊利用小鼠細胞與取自 Genotype-Tissue Expression (GTEx) Project 的人類腦組織作為樣本,同時與 Drop-Seq、sNuc-Seq 與其他低通量 scRNAseq 技術相行比較,發現 DroNc-Seq 的定序結果皆與先前研究結果一致,且能鑑別神經元、神經膠細胞與其他腦組織罕見細胞特有的表達特徵,以及區分密切相關的細胞亞型,不論是檢測的可靠度、靈敏度、規模或成本,皆具有高度競爭力。
開啟疾病研究的新頁
第一作者 Naomi Habib 表示, DroNc-Seq 可用於檢測人體細胞亞群極微小的差異,並幫助研究者發現更多未知的稀有細胞,對於神經退化性疾病如阿茲海默症、癌症及正常腦部老化過程的研究皆有應用價值。此外,通訊作者之一的 Aviv Regev 已著手規劃建立人類細胞圖譜 (Human Cell Atlas), DroNc-Seq 將可能成為此計畫主要應用的技術之一。
張鋒團隊已就 DroNc-Seq 的技術內容申請專利,且有數間公司表態未來授權的意願,開發單細胞定序應用平台的英國生技公司 Dolomite Bio 將於近期發表 DroNc-Seq 的檢測晶片,其微滴設計更小,而可增加高通量細胞核 RNA 定序的效能、成本更低,雖然總RNA 捕捉效率低於單細胞定序技術,卻能提供更完整的細胞核 RNA 及 pre-mRNA 表達訊息。
單細胞定序於 2013 年被《Nature Method》列為年度技術,隨著該技術的普及與成熟,研究者得更精確地由細胞層次探討生物體中各種細胞種系的演化親緣關係,在腫瘤基因體學、神經醫學或發育生物學等研究領域皆開始發揮強大的應用潛力,根據美國透明市場研究公司 (Transparency Market Research) 的報告顯示,全球轉錄體技術市場將於 2019 年達到 46 億美金。過去的疾病研究大多聚焦在分子機制之探討,期待透過高通量的單細胞核 RNA 定序技術,未來能以生態系的觀點看待細胞與疾病之間的關係,開啟臨床醫學研究的新紀元。
文 / Angela Chang
資料來源:
1. https://www.genomeweb.com/sequencing/advances-single-cell-rna-sequencing-enable-analysis-nuclei-higher-throughput
2, http://www.nature.com/nmeth/journal/v11/n1/full/nmeth.2801.html
3. https://www.broadinstitute.org/news/dronc-seq-microfluidic-single-nucleus-rna-sequencing
4. http://www.genengnews.com/gen-news-highlights/single-nucleus-rna-seq-merges-with-microfluidics/81254868
5. https://www.broadinstitute.org/blog/divide-and-conquer-new-single-cell-approach-broadens-range-cell-types-can-be-studied-brain
6. Habib, N. et al. Nature Methods (2017) doi:10.1038/nmeth.4407
7. Habib, N. et al. Science 353: 925–928 (2016).
©www.geneonline.news. All rights reserved. 基因線上版權所有 未經授權不得轉載。合作請聯繫:service@geneonlineasia.com






