2016 年,时任美国哈佛大学的谢晓亮(X.Sunney Xie)教授和亿康基因(Yikon Genomics)的陆思嘉(Sijia Lu)博士研究团队在《美国国家科学院院刊》(Proceedings of the National Academy of Sciences of the United States of America, PNAS USA)发表了一篇研究论文,透过一种自主开发名为 MALBAC(Multiple Annealing and Looping-based Amplification Cycles)的专利技术方法(WO2012166425A2 WIPO PCT,2012 年同步发表于 Science 杂志,序列扩增覆蓋效率高达 93%)进行全基因染色体扩增(Whole Genome Amplification)。
扩增技术优化!仅透过细胞培养液也能取得足量 cfDNA 进行胚胎染色体分析
该技术成功从胚胎细胞培养液中纯化扩增出悬浮的细胞游离 DNA(cfDNA),经过次世代基因定序分析(Next Generation Sequencing, NGS),对试管婴儿疗程的受精胚胎进行全染色体序列筛检,透过非侵入性的采样方式(亦不需在胚胎上打洞让囊胚腔液流出与细胞培养液混合),同样达成胚胎着床前染色体基因筛检(PGS/PGT-A)的目的。
研究数据显示,透过胚胎细胞培养液得到的基因定序结果与直接采取整颗胚胎细胞进行基因定序的黄金标准结果相比较,敏感度(Sensitivity)为 88.2%,特异性(Specificity)为 84.0%,阳性预测率(PPV)为 78.9%,阴性预测率为 91.3%,二者之间的吻合度达 85.7%,参与试验的 42 颗胚胎也都未有检测失败的情况,具有相当高的临床参考意义,开始受到全球生殖医学领域专家的高度关注。
细胞培养液混合囊胚腔液,试图取得较多的游离 DNA(cfDNA) : mi-PGS/mi-PGT-A
2017 至 2018 年间也有中国的研究团队尝试采用胚胎囊胚液(Blastocoel Fluid)与细胞培养液混合 embryo culture medium/blastocoel fluid(ECB)的取样方式,以获取较多游离 DNA,经过扩增与基因定序分析,与细胞采样的 PGT-A 和经过细胞采样的剩余胚胎的分析分别来做对照比较,并将这样的方法称为微侵入性胚胎着床前染色体基因筛检(mi-PGS/mi-PGT-A)。结果显示,若将剩余的整颗胚胎当作黄金标准对照组:
细胞采样 PGT-A 组
- 敏感度(Sensitivity)为 89.47%
- 特异性(Specificity)为 73.63%
- PGT-A 组和整颗胚胎组的结果一致性为 72.5%(27.5% 不同)
ECB 组
- 敏感度(Sensitivity)为 89.47%
- 特异性(Specificity)仅有 68.42%
- ECB 组和整颗胚胎组的结果一致性为 50%
最后,PGT-A 和 ECB 组的结果一致性则仅有 45%。
2018 年加拿大多伦多大学的研究团队也发表了相同方法学的研究论文,用胚胎囊胚液(Blastocoel Fluid)与细胞培养液混合 embryo culture medium/blastocoel fluid 发现:
- NI-PGT-A 与整颗剩余胚胎的一致性为 96.4%
- TE 细胞 PGT-A 和整颗剩余胚胎的一致性为 91.7%
最后,NI-PGT-A 与 TE 细胞 PGT-A 的一致性是 87.5%。
2019 年以陆思嘉(Sijia Lu)博士为首的研究团队则搜集了 41 颗胚胎来自 22 对亲代具有染色体转位的夫妻,以胚胎囊胚液(Blastocoel Fluid)与细胞培养液混合的方式并且将实验流程条件做了一些优化(BCM 组),与 TE 细胞采样组和剩余的整颗胚胎(BE 组)来做比较,不仅分析染色体倍数异常的结果(PGT-A),更进一步去分析这些胚胎发生染色体转位的情况(PGT-SR)。
PGT-A 的分析结果:
- BCM 组与 BE 组的一致性为 76.19%
- TE 组与 BE 组的结果一致性也为 76.19%
但令人意外的是,PGT-SR 的分析结果,BCM 组与 BE 组的一致性为 90.24%,TE 组与 BE 组的结果一致性则是 100%。
非侵入性的 NI-PGS/ni-PGT-A 结果,比传统胚胎切片的 PGS/PGT-A 更具代表性
2019 年美国哈佛大学的谢晓亮(X.Sunney Xie)教授团队和哈佛大学医学院旗下布莱根妇女医院(Brigham and Women’s Hospital)的权威胚胎学家 Catherine Racowsky 教授团队合作,再度在《美国国家科学院院刊》发表了关键性的研究成果:透过高效率的 MALBAC(Multiple Annealing and Looping-based Amplification Cycles)扩增技术不仅能直接采取胚胎细胞培养液中的 cfDNA 进行染色体分析,检测结果竟比传统采取外胚层细胞(Trophectoderm cell)的 PGS/PGT-A 更吻合整颗胚胎的真实染色体分布情况。
他们对于同一批胚胎分别采取胚胎细胞培养液、滋养外胚层细胞(Trophectoderm cell)和整颗胚胎(内胚层+滋养外胚层)三种检体样本进行扩增与基因定序分析,将整颗胚胎(内胚层+滋养外胚层)的结果当作黄金标准,分别用胚胎细胞培养液和滋养外胚层细胞(Trophectoderm cell)这两组的结果分别与整颗胚胎(黄金标准)做比较。
同时,他们发现将判定标准(Threshold)设定在 60% 细胞镶嵌(60% Mosaicism)讯号时,能够最精准区别的染色体整倍体(Euploidy)与染色体非整倍体(Aneuploidy)的胚胎结果判定。
在这样的判定标准制定下,二组的敏感度(Sensitivity)与阴性预测率(Negative Predictive Value, NPV)皆达到 100%,伪阳性率(FNR)则为 0%,几乎达到相同水平。更值得一提的是,NI-PGT-A 的组别在特异性(Specificity)80% 和阳性预测率(PPV) 91.7% 的表现都明显比传统 PGT-A 的 50% 和 78% 来得优异,伪阳性率(FPR)的表现 NI-PGT-A( 20.0%)也显著优于 PGT-A(50%),大幅改善过往 PGT-A 被反对者质疑的伪阳性率高所造成的道德伦理议题。
进一步深入分析二组分别在染色体非整倍体(Aneuploidy)和染色体片段缺失/重复(Deletion/Duplication/CNV)的表现,NI-PGTA 在非整倍体的结果的一致性(Concordance)达 93.8%,传统 PGT-A 的吻合度则为 82.0%,差异为 11.8%。而在染色体片段缺失/重复(Deletion/Duplication/CNV)的吻合度,NI-PGT-A 为 83.3%,传统 PGT-A 则为 62.0%,二组之间差异高达 21.3% 更为显著。
上述研究数据揭示著,NI-PGT-A 可能比传统 PGT-A 更能忠实呈现胚胎整体的染色体状况,尤其是在以往侦测较困难的染色体片段缺失/重复(Deletion/Duplication/CNV)项目,NI-PGA 的表现似乎更胜于传统 PGT-A,能提供更可靠的临床参考意义。
2020 年西班牙 Carlos Simons 研究团队也在《美国妇产科杂志》American Journal of Obstetrics and Gynecology 发表了他们在欧洲的研究,比较采取胚胎细胞培养液 cfDNA 和采取 TE 细胞进行胚胎染色体基因筛检的。研究数据显示 NI-PGT-A 和 TE biopsy PGT-A 二种不同采检方法的比较:
- NI-PGT-A 和 TE biopsy PGT-A(对照组)的一致性(Concordance)为 78.2%(全染色体结果接吻合 67.7% 部分吻合 10.5%)
- 敏感度(Sensitivity)为 81.7%
- 特异性(Specificity)为 77.4%
- 阳性预测率(PPV)为 75.0%
- 阴性预测率(Negative Predictive Value, NPV)为 83.6%
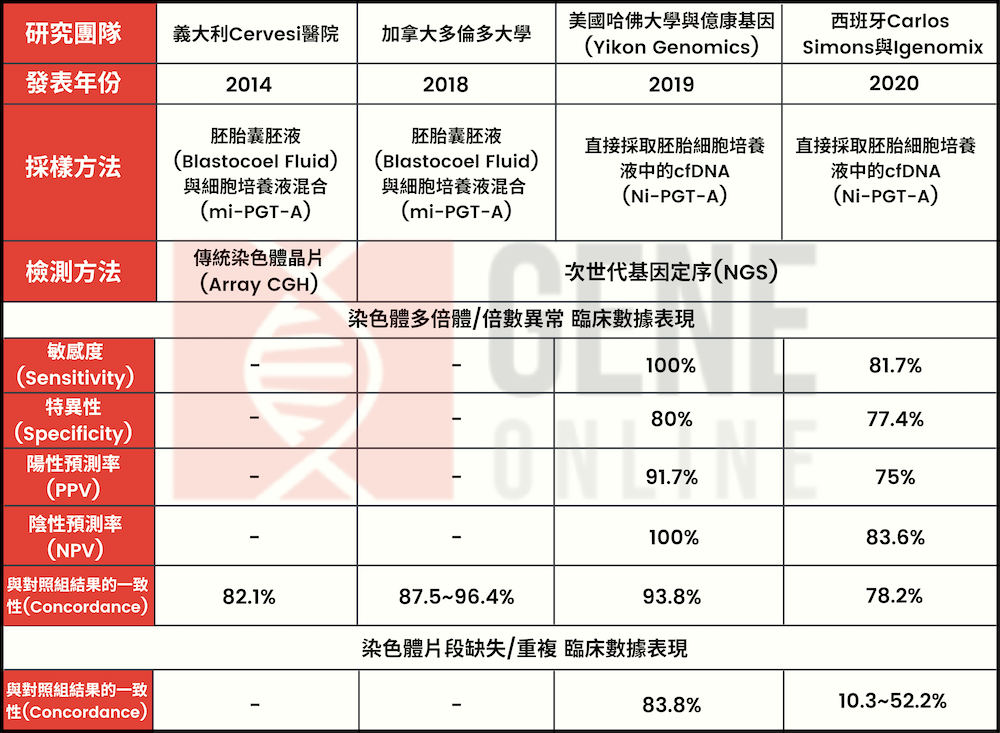
除了染色体倍数异常以外,NI-PGT-A 在研究当中也被用来检测染色体片段缺失/重复与 PGT-A 的吻合度,其研究结果显示,在染色体缺失的吻合度约为 52.2%,而在染色体重复的吻合度则仅有 10.3%,都明显低于染色体倍数异常的水准(P<0.0001)。
另一方面,此篇研究收案的检体当中,有 5.4% 的 NI-PGT-A 检体在 DNA 扩增与基因定序的过程中无法出具分析结果,相较于 PGT-A 的检测失败率 2.0%,检测失败的比例高出 2.7 倍,这样的结果更突显出稳定而高效能的 DNA 扩增技术在 NI-PGT-A 检测当中扮演至关成败的关键性角色。
由于细胞培养液中的 DNA 含量非常稀少,精良的全染色体扩增(WGA)技术和胚胎培养与检体的采集方法就变得至关重要,从后续的研究当中也陆续发现证实,影响 NI-PGS/NI-PGT-A 分析结果的二大关键因素:
过低的 cfDNA 浓度/含量或扩增效率与品质不够好的全基因染色体扩增技术
NI-PGS/NI-PGT-A 的技术原理和近年火红的非侵入性胎儿染色体基因检测(NIFTY/NIPT)有着异曲同工之妙,都是从液态生物检体(Liquid Biopsy)中萃取纯化出游离的 DNA 片段后续进行全基因染色体扩增和 DNA 基因定序分析。然而,2018 年美国妇产科医学权威 Diana Bianchi 教授发表在新英格兰杂志的 review 文章提到,已有研究文献证实过低的胎儿 cfDNA 浓度会影响是导致 NIPT 分析结果伪阴性最主要的原因之一。
因此,在部分 NI-PGS/NI-PGT-A 的研究论文当中也有提到,如何取得足量的 cfDNA 和扩增效率与品质优良的全基因染色体扩增(WGA)流程,是影响 NI-PGS/NI-PGT-A 分析结果的最关键性因素。
胚胎上来自母方/父方来源的生物性因素
胚胎受精早期卵子上残留来自母体 DNA 或是来自黏着在卵子表面的残留精虫 DNA,都可能会在分析过程中造成极大的干扰因素,因此在检体采集的流程中都会要求在胚胎受精后的第 3 或 4 天进行更换胚胎培养液甚至润洗的步骤,避免这些干扰因素残留在胚胎细胞培养液中影响后续 NI-PGS/NI-PGT-A 分析结果。
攸关 NI-PGS 检测品质与成效的决定性因素
同理可证,来自胎儿和胚胎的 DNA 样本都极其微量,对于检测技术、实验流程与环境控管都具有极高的挑战性。cfDNA 是游离在细胞培养液当中的破碎 DNA 片段,非常容易受到外在环境条件的影响而被降解而破坏殆尽,相较 PGT-A 是直接取得 3-5 颗 TE 细胞进行 DNA 萃取与扩增,ni-PGT-A 需要更高的技术水平和更严格的条件控管,以下可能是攸关检测结果品质的三大关键因素:
高效且均匀的 DNA 扩增技术
来自胚胎细胞 cfDNA 非常微量,必须经过全基因染色体扩增(Whole Genome Amplification)的关键步骤来等量放大所有的cfDNA片段,才能够忠实地反映出从胚胎染色体 DNA 拷贝数变异(Copy Number Variation, CNV)的情况。因此,必须采取高效且均匀的DNA扩增技术,例如: MALBAC(Multiple Annealing and Looping-based Amplification Cycles),来解决检测失败率偏高和因为扩增不均匀而造成基因定序分析的二大关键性问题。
无尘室规格的全正负压实验室操作环境
NI-PGS/NI-PGT-A 检测所采取的胚胎 cfDNA,由于是极短片段(100bp左右)的微量DNA,非常容易受到外在因素而降解殆尽,直接导致检测失败。操作环境中只要有任何一点不明物质存在,都可能造成 DNA 样本受到污染,会直接影响 NI-PGS/NI-PGT-A 的基因定序分析精准度。
因此,全程必须要在具备无尘室等级的全正负压实验室进行操作,才能确保检测品质。若仅在一般的分子生物实验室或没有全正负压的无尘实验室环境操作,虽然成本相对低廉,但可能必须承担更多的检测失败和品质风险。
是否具有公开临床数据验证的技术来源
NI-PGS/NI-PGT-A 是一项崭新的新技术,目前已经知道有包括: 意大利 Cervesi 医院、加拿大多伦多大学、西班牙团队和美国哈佛大学谢晓亮教授技术授权的亿康基因(Yikon Genomics)等研究团队都有将他们的技术与研究数据公开发表,不同研究团队得到的临床数据和效度也因为检测技术、演算方法、样本人种等因素而有所不同。
现在也有一些检测服务供应商,陆续开始在欧、美、亚洲等地提供相关的检测服务。然而,在进行临床检测服务之前,必须先厘清其技术来源,是否已具有临床数据公开发表可供验证,甚至考量是否有足够在地亚洲人种样本做为数据比对分析基础,以确保 NI-PGS/NI-PGT-A 检测分析的可信赖度。
此外,更应该避免依样画葫芦仿傚模式,在未经公开验证与临床检测效度不明的情况下贸然提供相关检测的临床服务,避免衍生无法掌控的风险和争议。
非侵入性胚胎着床前染色体筛检(NI-PGS/PGS)临床应用的挑战与展望
NI-PGS/NI-PGT-A 的原理是蒐集胚胎释放到细胞培养液中的 cfDNA,然而在胚胎受精后一般常见的群体培养(Group Culture)是将一批数颗胚胎同时培养在同一个培养液滴或槽内,因为共用培养液而无法确保 DNA 的来源性,造成分析上的困难。
这个情况与非侵入性胎儿染色体基因检测(NIFTY/NIPT)在多胞胎妊娠案例上所遇到的困境限制非常类似。因此接受检测的胚胎必须采取单独培养(Single Culture),让每一个胚胎在个别独立的培养槽或培养液滴当中培养生长,才能蒐集到单一来源性的 DNA。
另一方面,近年来蔚为风尚的 3.5 代试管婴儿,是在 IVF 疗程搭配 PGS/PGT-A 再加上缩时摄影胚胎培养(Time Lapse Embryo Culture System)的即时记录监测,甚至运用影像分析软件的算法来辅助判断胚胎的优劣等级(但从外观仍无法精准分辨胚胎是否有染色体异常,仍须透过基因检测分析来找出染色体异常的胚胎),是目前临床上相当受到欢迎的试管婴儿疗程配套方案。
但是由于大多的缩时摄影胚胎培养(Time Lapse Embryo Culture System)的设计是在数个胚胎孔槽之间相通共用细胞培养液,无法满足胚胎需要在单独培养液环境中的条件,成为目前 NI-PGS/NI-PGT-A 尚无法大量应用的技术障碍之一。
不过,正因为 NI-PGS/NI-PGT-A 强烈的市场需求潜力,已有部份的缩时摄影胚胎培养(Time Lapse Embryo Culture System)设备业者开始投入设计可符合独立培养条件的胚胎培养盘,相信在之后这样的技术问题将可能迎刃而解。可以预期不久的未来,这个崭新的技术将会在生殖医学领域有非常广泛且重要的应用。
作者:John Hung
生殖医学崭新亮点(上)- 非侵入性胚胎着床前染色体基因筛检(Ni-PGT-A)参考资料:
- Genomic DNA in human blastocoele fluid. Reprod Biomed Online. 2013 Jun;26(6):603-10.
- Blastocentesis: a source of DNA for preimplantation genetic testing. Results from a pilot study. Fertil Steril. 2014 Dec;102(6):1692-9.e6.
- Noninvasive chromosome screening of human embryos by genome sequencing of embryo culture medium for in vitro fertilization. Proc Natl Acad Sci 2016; 113:11907–11912.
- Non-invasive preimplantation aneuploidy screening and diagnosis of beta thalassemia IVSII654 mutation using spent embryo culture medium. Ann Med 2017; 49:319–328.
- Preimplantation genetic screening with spent culture medium/blastocoel fluid for in vitro fertilization. Sci Rep 2018;8: 9265.
- Minimally invasive preimplantation genetic testing using blastocyst culture medium. Hum Reprod 2019; 34:1369–1379.
- Chromosome screening using culture medium of embryos fertilised in vitro: A pilot clinical study. J Transl Med 2019; 17:1–8.
- Noninvasive preimplantation genetic testing for aneuploidy in spent medium may be more reliable than trophectoderm biopsy. Proc Natl Acad Sci 2019; 116:201907472.
- Multicenter prospective study of concordance between embryonic cell-free DNA and trophectoderm biopsies from 1301 human blastocysts. J Obstet Gynecol. 2020 Nov;223(5):751.e1-751.e13.
- Evaluation of a novel non-invasive preimplantation genetic screening approach. PLoS One. 2018; 13(5): e0197262.
- Sequencing of Circulating Cell-free DNA during Pregnancy. N Engl J Med. 2018 Aug 2;379(5):464-473.
©www.geneonline.news. All rights reserved. 基因线上版权所有 未经授权不得转载。合作请联系:service@geneonlineasia.com